Redacción Farmacosalud.com
Un trabajo liderado por la Universidad Pompeu Fabra de Barceona (UPF) explica un nuevo método computacional que permite describir las alteraciones de splicing* en nueve tipos de cáncer utilizando datos de secuenciación de ARN. Mediante este nuevo enfoque metodológico, se ha descubierto que en la enfermedad cancerosa hay diferentes interruptores de splicing para un mismo tipo de cáncer en diferentes pacientes, y muestra que estos interruptores se pueden identificar con alta precisión, tanto en los tumores como en las muestras control sin tumor, así como también entre tipos de tumor diferentes, proporcionando una valiosa firma predictiva de la enfermedad. Según han indicado a www.farmacosalud.com fuentes de la UPF, los nueve tipos de cáncer en los que se han descrito alteraciones de splicing son: mama, colon, cabeza y cuello, dos tipos de tumor renal, dos tipos de tumor pulmonar, próstata y tiroides.
Fuente: UPF
Durante muchos años, los científicos han luchado por entender y curar el cáncer. El estudio del genoma de muchos de los diferentes tumores identificados ha sido fundamental para detectar alteraciones recurrentes en varios tipos de cáncer, ha facilitado su clasificación y el desarrollo de nuevas estrategias terapéuticas. La mayor parte de las estrategias utilizadas en proyectos de genoma del cáncer se basan en la búsqueda de alteraciones genéticas o cambios en la expresión de genes, hallazgos aún insuficientes para entender completamente el proceso tumoral, prescribir los mejores tratamientos o mejorar el diagnóstico de la enfermedad. De ahí que sea primordial encontrar nuevas firmas de cáncer. Cada día hay más y más evidencia de que las alteraciones en el programa de regulación del splicing juegan un papel importante en la transformación tumoral. Un trabajo liderado por Eduardo Eyras, investigador ICREA del Departamento de Ciencias Experimentales y de la Salud (CEXS) de la UPF, así lo demuestra.
Una nueva y valiosa firma predictiva del cáncer
El trabajo se ha publicado en la revista ‘Nucleic Acids Research’ y explica los resultados de aplicar un nuevo método computacional desarrollado por estos científicos a los datos del proyecto Atlas del Genoma del Cáncer (TCGA), la rama financiada por los Institutos Nacionales de Salud (NIH) de los EEUU, del proyecto ICGC (International Cancer Genome Consortium). Este trabajo representa el primer análisis a gran escala publicado que describe las alteraciones de splicing en nueve tipos de cáncer utilizando datos de secuenciación de ARN a partir de más de 4.000 muestras.
Mediante este nuevo enfoque metodológico, se han descubierto datos relevantes para el futuro manejo del cáncer. Se ha descubierto que en la enfermedad cancerosa hay diferentes interruptores de splicing para un mismo tipo de cáncer en diferentes pacientes, y muestra que estos interruptores se pueden identificar con alta precisión, tanto en los tumores como en las muestras control sin tumor, así como también entre tipos de tumor diferentes, proporcionando una valiosa firma predictiva de la enfermedad. En particular, el método proporciona reglas simples basadas en la expresión de algunas moléculas de ARN que potencialmente permitirían la determinación del tipo de cáncer, a partir de una muestra de ARN del paciente.
Los cambios de splicing o interruptores pueden alertar sobre un cáncer
Por ejemplo, este mismo trabajo ha identificado firmas para el subtipo de cáncer de mama triple negativo, uno de los subtipos más agresivos de tumor mamario. En general, este nuevo método de cálculo computacional revela nuevas firmas de cáncer en términos de isoformas de ARN expresados específicamente en los tumores, proporcionando potenciales nuevas dianas moleculares para el pronóstico y el tratamiento. Por lo tanto, aporta nuevas alteraciones moleculares para la caracterización de tumores individuales.
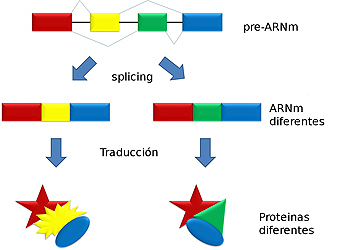
Splicing
Fuente: UPF
El splicing* (corte y unión) es un proceso que tiene lugar en el núcleo de la célula mediante el cual una molécula larga de ARN, transcrita a partir del genoma, se procesa para eliminar segmentos llamados intrones, dando lugar a un ARN transcrito. Por splicing alternativo se entiende un mecanismo por el cual se generan múltiples transcritos de ARN del mismo gen mediante la eliminación de intrones, y de diferentes maneras. Este proceso está estrechamente regulado y da lugar a proteínas con funciones específicas de tipo celular, de activación o de inhibición de determinadas funciones, o incluso puede dar lugar a una forma de activar o desactivar la función de algunos genes. Este cambio en el rol del splicing se conoce como interruptor de splicing. Los cambios de splicing o interruptores que no están directamente regulados por la célula pueden dar estados alterados celulares y conducir hacia una enfermedad. Por lo tanto, la determinación de las alteraciones de corte y unión o splicing alternativo en los tumores puede ser fundamental para el desarrollo de dianas moleculares específicas de tumor que pueden ayudar en el pronóstico y en el tratamiento posterior. Sin embargo, el estudio del splicing en el cáncer se ve obstaculizada por la heterogeneidad que presentan los tumores similares en diferentes individuos, además de la variabilidad normal existentes entre individuos.
Método computacional compatible con la variabilidad biológica y técnica
La aportación relevante de este trabajo que acaban de publicar Sebestyén, et al., ha sido el descubrimiento de un nuevo método computacional, compatible con la variabilidad biológica y técnica, que identifica los interruptores de splicing en un gran número de muestras tumorales con una alta precisión en los conjuntos de datos de los que se dispone. Además, este método es capaz de identificar cambios complejos de splicing alternativo que no se podrían describir utilizando metodologías estándar. Además, el nuevo método es independiente de otros parámetros a tener en cuenta, como que el análisis de los datos de secuenciación de ARN provenga de muestras de varios laboratorios y plataformas tecnológicas. Eyras ha contado con la colaboración de Endre Sebestyén, miembro de su grupo de investigación en Genómica Computacional, así como de la colaboración especial de Michal Zawisza, informático de la Universidad Politécnica de Cataluña, recién graduado y que a raíz de este trabajo ha mostrado tener un gran interés y disposición para la biología, ha informado la UPF mediante un comunicado.
Trabajo de referencia
Endre Sebestyén, Michał Zawisza , Eduardo Eyras (2015), 'Detection of recurrente alternative splicing switches in tumor samples Reveal novela firmas of cancer', Nucleic Acids Research, doi: 10.1093 / NAR / gku1392.




